Резюме
PGC-1α (пероксисома активированный распространителями рецептор γ коактиватор 1α) является важным регулятором митохондриальной биогенетики (синтеза, обновления и роста митохондрий) и основным регулятором ферментов, вовлеченных в окислительное фосфорилирование. Недавние находки продемонстрировали, что полиморфизм нуклеотида (SNP) Gly482Ser в PGC-1α гене влияет на чувствительность к инсулину, метаболизм жиров в крови и связан с myocyte enhancer factor 2 (MEF2). У людей-носителей этого SNP, как показано, были сниженные сердечно-легочный fitness и более высокий риск заболеть диабетом 2 типа. Здесь, мы исследовали реакции нетренированных мужчин с Gly482Ser SNP к 10-недельной программе тренировок на выносливость (циклическая, 3 раза по 60 минут в неделя, частота сердцебиения относительно VO2peak — 70-90%). Количественные данные из анализа биопсий из vastus lateralis мышцы показали, что группа SNP, в отличие от контрольной группы, не показала адаптации к тренировкам в виде увеличения содержания медленных (окислительных) волокон. Капилляризация, митохондриальная плотность, митохондриальные ферменты и внутримиоцеллюларное содержание жиров увеличились так же в обеих группах. Эти результаты указывают, что сниженное связывание MEF2 с PGC-1α у людей с этим SNP препятствует преобразованию мышечного волокна в медленное под воздействием тренировок.

Мышечная композиция и капилляризация
*картинка показывает на A количество медленных, на B количество быстрых, на C капилляры
Пример срезов биопсии vastus lateralis мышцы, имунноокрашенных для
(A) волокон с медленным (slow) миозином (MHC),
(B) быстрых (fast) MHC и
(C) маркер CD31 эндотелиальной клетки (капилляризация). Числа 1, 2 и 3 отмечают полностью идентичные медленные мышечные волокна в срезе. Шкала масштаба: 50 μm.
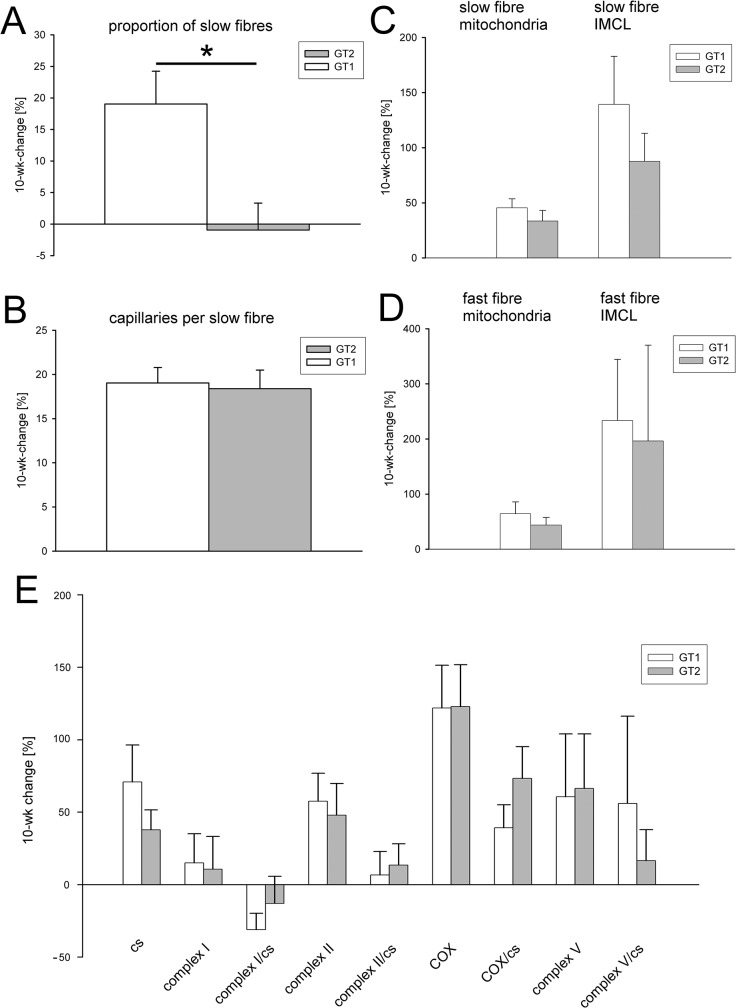
Относительные изменения [в %] особенностей мышцы в ответ на 10ти недельную тренировку выносливости.(A) Группа контроля (GT1) показывает значительное увеличение пропорции медленных мышц после тренировки, в то время как носители генов SNP Gly482Ser (GT2) нечувствительны к тренировкам. (B) нет никакого различия между GT1 и GT2 в увеличении капилляров. (C) Контрольные группы (GT1) и носители SNP (GT2) не показывают существенных различий в вызванных тренировками увеличении объемной плотности митохондрии в медленных мышечных волокнах и внутримиоцеллюларного липида (IMCL). (D) увеличение митохондрий в быстрых мышечных волокнах и IMCL также подобны в GT1 и GT2. (E) Точно так же послетренировочное увеличения количества митохондрий, выраженного в активности CS и OXPHOS не имеют значимых различий между GT1 и GT2. GT1 (n = 13), GT2 (n = 15); различия между группами p <0.05.
Исходные параметры генотипов
| Группы | Всего | GT1 (контрольная) | GT2 (исследуемая) | |
|---|---|---|---|---|
| SNP-PPARGC1A | G/A, G/A, A/A | G/G (Gly/Gly) | X/A (X/Ser) | p |
| Sample size, n | 28 | 13 | 15 | |
| Возраст (yrs) | 58.7 ± 1.2 | 59.3 ± 2.1 | 58.1 ± 1.5 | ns |
| BM (kg) | 88.0 ± 2.2 | 89.9 ± 4.2 | 86.3 ± 2.0 | ns |
| BMI (kg m-2) | 27.8 ± 0.8 | 28.2 ± 1.4 | 27.4 ± 3.1 | ns |
| Waist (cm) | 101.0 ± 1.9 | 101.7 ± 3.5 | 100.4 ± 2.0 | ns |
| SF proportion (%) | 55.5 ± 2.1 | 50.5 ± 2.5 | 59.9 ± 2.8 | 0.02 |
| HF proportion (%) | 1.5 ± 0.2 | 1.5 ± 0.3 | 1.4 ± 0.3 | ns |
| Cap/SF | 6.50 ± 0.13 | 6.37 ± 0.15 | 6.61 ± 0.20 | ns |
| Mito-SF (%) | 7.47 ± 0.25 | 6.97 ± 0.36 | 7.90 ± 0.33 | ns |
| Mito-FF (%) | 3.53 ± 0.16 | 3.35 ± 0.19 | 3.67 ± 0.25 | ns |
| Lip-SF (%) | 1.19 ± 0.13 | 1.32 ± 0.21 | 1.07 ± 0.16 | ns |
| Lip-FF (%) | 0.39 ± 0.05 | 0.41 ± 0.10 | 0.37 ± 0.05 | ns |
| mtDNA copies/cell | 4212 ± 355 | 4095 ± 401 | 4313 ± 578 | ns |
| CS [mU/mg prot.] | 251 ± 17 | 259 ± 22 | 244 ± 26 | ns |
| C I [mU/mg prot.] | 39 ± 2 | 41 ± 3 | 36 ± 4 | ns |
| C II [mU/mg prot.] | 63 ± 5 | 63 ± 7 | 62 ± 8 | ns |
| COX [mU/mg prot.] | 280 ± 23 | 312 ± 35 | 252 ± 30 | ns |
| C V [mU/mg prot.] | 84 ± 10 | 89 ± 15 | 80 ± 15 | ns |
| Complex I/CS | 0.16 ± 0.01 | 0.17 ± 0.01 | 0.15 ± 0.01 | ns |
| Complex II/CS | 0.26 ± 0.02 | 0.25 ± 0.02 | 0.26 ± 0.03 | ns |
| COX/CS | 1.17 ± 0.09 | 1.27 ± 0.13 | 1.08 ± 0.12 | ns |
| Complex V/CS | 0.32 ± 0.03 | 0.31 ± 0.04 | 0.32 ± 0.05 | ns |
Values are means ±S.E. after one-way ANOVA; GT1 = major allele type in PPARGC1A (G/G, wild type), GT2 = homozygous and heterozygous for minor allele frequency in PPARGC1A (A/A, G/A); BM = body mass, BMI = body mass index; VO2-RCP = oxygen uptake at respiratory compensation point (RCP, second ventilatory threshold, aerobic capacity), VO2-peak = peak oxygen uptake at cessation (aerobic power), PRCP = mechanical power at RCP in Watt, Pmax = maximum mechanical power in Watt; SF = slow fibre (MHC slow+/MHC fast-); HF = slow-fast hybrid fibre (MHC slow+/MHC fast+); cap/SF = capillaries per slow fibre; Mito-SF = mitochondria in slow fibre; Mito-FF = mitochondria in fast fibre; Lip-SF = lipid droplets in slow fibre; Lip-FF = lipid droplets in fast fibre; mtDNA = mitochondrial DNA; CS = citrate synthase; C I—V = complex I—V; COX = cytochrome c oxidase; p = significance level between the genotype (GT) groups; ns = not significant.
Результаты генотипов после 10ти недельных тренировок на велосипеде и антропометрические данные, аэробная производительность, сила, ферменты митохондрий и проч.
| Groups | Total | GT1 | GT2 | |
|---|---|---|---|---|
| SNP-PPARGC1A | G/G (Gly/Gly) | A/A (X/Ser) | p | |
| n | 28 | 13 | 15 | |
| BM (kg) | -1.3 ± 0.3*** | -1.2 ± 0.5* | -1.3 ± 0.5* | ns |
| BMI (kg m-2) | -0.4 ± 0.1*** | -0.4 ± 0.1* | -0.4 ± 0.2* | ns |
| Waist (cm) | -0.7 ± 0.5 | -1.3 ± 0.8 | -0.2 ± 0.4 | ns |
| SF proportion (%) | 3.3 ± 2.1 | 8.9 ± 2.6** | -1.5 ± 2.6 | 0.01 |
| HF proportion (%) | -0.4 ± 0.3 | -0.6 ± 0.5 | -0.2 ± 0.3 | ns |
| Cap/SF | 1.18 ± 0.07*** | 1.19 ± 0.10*** | 1.17 ± 0.10*** | ns |
| Mito-SF (%) | 2.71 ± 0.42*** | 2.99 ± 0.50*** | 2.46 ± 0.66** | ns |
| Mito-FF (%) | 1.73 ± 0.32*** | 2.35 ± 0.47*** | 1.24 ± 0.41** | ns |
| Lip-SF (%) | 0.89 ± 0.19*** | 1.14 ± 0.26*** | 0.68 ± 0.28* | ns |
| Lip-FF (%) | 0.10 ± 0.09 | 0.19 ± 0.15 | 0.02 ± 0.11 | ns |
| mtDNA copies/cell | 122 ± 377 | 90 ± 323 | 149 ± 658 | ns |
| CS [mU/mg prot.] | 108 ± 28*** | 152 ± 48** | 71 ± 31* | ns |
| C I [mU/mg prot.] | -0.1 ± 5 | 1.5 ± 8 | -1.6 ± 7 | ns |
| C II [mU/mg prot.] | 21 ± 7** | 26 ± 9** | 16 ± 10 | ns |
| COX [mU/mg prot.] | 294 ± 54*** | 356 ± 89** | 240 ± 64** | ns |
| C V [mU/mg prot.] | 43 ± 25 | 50 ± 34 | 37 ± 37 | ns |
| Complex I/CS | -0.05 ± 0.02** | -0.06 ± 0.02** | -0.05 ± 0.02 | ns |
| Complex II/CS | 0.00 ± 0.02 | -0.00 ± 0.03 | -0.00 ± 0.03 | ns |
| COX/CS | 0.44 ± 0.12*** | 0.36 ± 0.14* | 0.51 ± 0.19* | ns |
| Complex V/CS | 0.00 ± 0.05 | 0.00 ± 0.06 | -0.01 ± 0.08 | ns |
Values are paired differences ± SE from paired sample t-Test after testing with general linear model for repeated measures; p = significant interaction between time and genotype; *p≤.05, **p≤.01 and ***p ≤.001 indicate significant differences of pre vs. post measures within group; ns, not significant; for other abbreviations see Table 1.
2015 апрель; 10 (4): e0123881. doi: 10.1371/journal.pone.0123881. eCollection 2015..Steinbacher P1, Feichtinger RG2, Кеденко L3, Кеденко I3, Рейнхардт S1, Schönauer AL1, Leitner I1, Sänger AM1, Stoiber W1, Kofler B2, Förster H4, Paulweber B3, Ring-Dimitriou S5.
